
이번에 알아볼 것은 차원축소 알고리즘의 대표적인 PCA(주성분 분석)에 대해서 알아보겠습니다.
Tabular type의 데이터에서 차원을 축소한다는 것은 곧, 변수의 개수(또는 피처의 개수)를 줄인다는 것을 의미하는데요.
너무 많은 피처들이 있을때 이들을 줄이는 것은 어떤 의미이며, 왜 해야되는지 등등에 대해서 PCA를 통해 알아보겠습니다.
1. PCA란
- PCA는 Principal Component Analysis의 약자로 주성분 분석으로 불립니다.
- 이는 다차원의 데이터에서 차원을 줄이고 주요 정보를 추출하기 위한 통계적인 방법인데요
* 이때 차원은, 피처(feature) 혹은 변수(variable)의 숫자를 의미합니다.
* 차원이 높다는 것은 피처 혹은 변수가 많다는 것을 의미하며, 이는 예측하고자하는 타겟과 연관된 변수가 많음을 의미합니다.
- PCA는 데이터의 차언을 줄이면서 데이터의 분산을 최대한 보존하는, "주요성분"을 찾아내는데 사용됩니다.
- 이를 통해, 데이터의 시각화(다차원에서 인간이 인지가능한 3차원이하로 축소가 가능하기 때문), 특징 선택(혹은 변수 선택), 노이즈 제거, 차원 축소 등 다양한 분야에서 활용됩니다.
- PCA에 대한 주요 내용을 정리하면 다음과 같은데요
a) 정의
· PCA는 다차원 데이터의 주요 정보를 포함하는 새로운 축(주성분)을 찾아내는 방법입니다.
· 이러한 주성분들은 데이터의 분산을 최대한 보존하는 방향으로 정의됩니다.
· 주성분은 데이터의 공분산 행렬의 고유 벡터(eigenvector)들로 표현됩니다.
b)특징
· 주성분은 서로 직교(orthogonal)하며, 첫 번째 주성분은 데이터의 분산을 가장 많이 설명하고, 두 번째 주성분은 첫 번째 주성분과 직교하며 데이터의 다른 측면을 설명합니다.
· PCA는 데이터의 노이즈를 줄이고, 상관 관계가 있는 변수를 독립적으로 만들어줌으로써 데이터의 해석을 용이하게 해줍니다.
· 차원 축소를 통해 데이터의 시각화가 용이하며, 고차원 데이터의 경우 계산 비용을 줄일 수 있습니다.
c)활용시 이점:
· 주성분 분석은 데이터 압축과 차원 축소에 주로 사용됩니다. 예를 들어, 고차원 데이터를 2D 또는 3D로 시각화하여 데이터 패턴을 이해하기 쉽게 만들 수 있습니다.
· PCA는 특징 선택(feature selection)과 특징 추출(feature extraction)에 활용됩니다. 중요한 특징들을 강조하거나 관련 없는 특징을 제거함으로써 모델의 성능을 향상시킬 수 있습니다.
d)단점:
· PCA는 데이터의 선형 관계를 기반으로 주성분을 찾으므로 비선형 데이터에는 적용하기 어렵습니다. 이를 해결하기 위해 커널 PCA와 같은 확장된 기법들이 개발되었습니다.
· 주성분의 해석이 어려울 수 있습니다. 주성분은 기존 변수들의 선형 조합으로 표현되므로 어떤 의미를 가지는지 명확하지 않을 때가 있습니다.
- PCA를 하는 과정은 하는 아래와 같습니다.
1) 먼저, 데이터를 준비합니다.
2) 데이터를 표준화해줍니다. PCA도 선형 관계를 기반으로 주성분을 찾기에 데이터가 표준화되어 있을수록 그 퍼포먼스가 높아지므로 표준화 과정을 거칩니다.
* 표준화(Standardization) : 각 특성을 평균이 0, 표준편차가 1이 되도록 변환


3) 공분산 행렬을 계산해줍니다.
* PCA는 데이터의 분산을 최대로 보존하는 주성분을 찾는 방법입니다. 공분산(Covariance)행렬은 데이터의 분산과 각 특성 간의 상관관계를 나타내는데 사용되며, 이를 구하면 데이터 간의 선형 관계를분석하고 주성분을 추출하는데 도움이 됩니다.


4) 고유값(Eigenvalues)과 고유벡터(Eigenvectors)를 계산해줍니다. 이를 통해 주성분을 정의할 수 있는데요.
* 고유값은 주성분의 중요도를 나타냅니다. 고유값이 클수록 해당 주성분이 데이터의 분산을 많이 설명한다고보며, 고유값이 높은 순서대로 주성분1(PC1), 주성분2(PC2) 등의 순이 됩니다.
* 고유벡터는 주성분의 방향을 나타냅니다. 고유벡터는 서로 직교하며, 주성분의 방향을 나타내므로 데이터를 새로운 축으로 변환하는데 사용이 됩니다.


5) 주성분 계산


위 과정을 예제 데이터를 생성해서 시각화 해보면 아래와 같습니다.
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
|
import numpy as np
import matplotlib.pyplot as plt
from sklearn.preprocessing import StandardScaler
# 선형적인 데이터 생성: 두 번째 특성 방향으로 큰 분산
np.random.seed(0)
data = np.random.randn(1000, 2)
data[:, 0] = 2 * data[:, 1] # 두 번째 특성 방향으로 큰 분산 추가
# 노이즈 추가
noise = np.random.normal(0, 0.2, size=(1000, 2))
data += noise
# 2. 데이터 표준화
scaler = StandardScaler()
scaled_data = scaler.fit_transform(data)
# 3. 공분산 행렬 계산
cov_matrix = np.cov(scaled_data, rowvar=False)
# 4. 공분산 행렬의 고유값과 고유벡터 계산
eigenvalues, eigenvectors = np.linalg.eig(cov_matrix)
# 5. 주성분(PC) 계산
principal_components = scaled_data.dot(eigenvectors[:, 0]) # 첫 번째 주성분(PC1)
# PC1 방향을 나타내는 벡터
pc1_vector = eigenvectors[:, 0]
# PC1 시각화
plt.figure(figsize=(8, 6))
plt.scatter(scaled_data[:, 0], scaled_data[:, 1], c=principal_components, cmap='viridis')
plt.colorbar(label='PC1')
plt.quiver(0, 0, pc1_vector[0], pc1_vector[1], angles='xy', scale_units='xy', scale=2, color='red', label='PC1 Direction')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
plt.title('Visualization of PC1 with PC1 Direction (Noisy Data)')
plt.legend()
plt.show()
|

* 해당 데이터는 feature1 과 feature2, 이 2개의 feature를 가진 데이터입니다.
* 데이터의 분포를 봤을때, 선형적인 관계를 가지고 있음을 알 수 있고, 약 45도의 선이 해당 데이터의 가장 높은 분산을 나타내는 선인데요.
* 공분산을 활용해 이 벡터의 고유 벡터를 계산하면 해당 데이터를 가장 잘 설명할 수 있는(가장 분산이 큰 방향의) 벡터는 위에서 빨간색으로 표시한 PC1이 되겠습니다.
* 이를 정리해보면, 원 데이터는 Feature1과 Feature2로 이루어진 데이터이지만, 공분산 계산, 고유값 및 고유벡터 계산을 통해 주성분1을 도출할 수 있었고, 해당 주성분1 한 개만으로도 데이터가 어느정도 설명이 된다는 것을 의미합니다.
|
1
2
3
4
5
6
7
8
9
10
|
# 결과를 데이터프레임으로 정리
cov_matrix_df = pd.DataFrame(cov_matrix, columns=['Feature 1', 'Feature 2'], index=['Feature 1', 'Feature 2'])
eigenvalues_df = pd.DataFrame({'Eigenvalues': eigenvalues}, index=['PC1', 'PC2'])
eigenvectors_df = pd.DataFrame(eigenvectors, columns=['PC1', 'PC2'], index=['Feature 1', 'Feature 2'])
explained_variance_ratio_df = pd.DataFrame({'Explained Variance Ratio': explained_variance_ratio}, index=['PC1', 'PC2'])
# 출력
print("공분산 행렬:\n", cov_matrix_df)
print("\n고유값:\n", eigenvalues_df)
print("\n고유벡터:\n", eigenvectors_df)
|
cs |

* 이때, 데이터에 대한 주성분(PC)별 설명력은 Explanied Varaince를 통해서 정의됩니다. 즉, 각 주성분벡터가 이루는 축에 투영한 결과의 분산 비율을 말하며, 각 고유값(eigenvalue)의 비율과 같은 의미입니다.
|
1
2
3
|
# explained_variance_ratio 계산
explained_variance_ratio = eigenvalues / np.sum(eigenvalues)
print("\nExplained Variance Ratio:\n", explained_variance_ratio_df)
|
cs |

* 위 데이터의 PCA결과 PC1은 전체 데이터 분산의 98.77%를 설명할 수 있고, PC2가 그 나머지를 설명할 수 있다고 보시면 되겠습니다.
2. 파이썬을 활용한 PCA 구현 - iris 데이터 활용
이번에는 파이썬을 활용해 PCA를 구현해보고 결과를 도출해보겠습니다.
*먼저 분석에 사용할 iris 데이터와 필요 라이브러리를 불러옵니다.
|
1
2
3
4
5
6
7
8
9
10
11
12
13
|
from sklearn.datasets import load_iris
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
# 사이킷런 내장 데이터 셋 API 호출
iris = load_iris()
# 넘파이 데이터 셋을 Pandas DataFrame으로 변환
columns = ['sepal_length','sepal_width','petal_length','petal_width']
irisDF = pd.DataFrame(iris.data , columns=columns)
irisDF['target']=iris.target
irisDF.head(3)
|
cs |

* 각 클래스의 분포를 4개의 feature 중 sepal length와 sepal width만으로 표현해봅니다.
|
1
2
3
4
5
6
7
8
9
10
11
12
13
|
#setosa는 세모, versicolor는 네모, virginica는 동그라미로 표현
markers=['^', 's', 'o']
#setosa의 target 값은 0, versicolor는 1, virginica는 2. 각 target 별로 다른 shape으로 scatter plot
for i, marker in enumerate(markers):
x_axis_data = irisDF[irisDF['target']==i]['sepal_length']
y_axis_data = irisDF[irisDF['target']==i]['sepal_width']
plt.scatter(x_axis_data, y_axis_data, marker=marker,label=iris.target_names[i])
plt.legend()
plt.xlabel('sepal length')
plt.ylabel('sepal width')
plt.show()
|
cs |

4개의 피처 중 2개의 피처로만 나타내보니 versicolor와 virginica는 서로 구분이 잘 안됩니다.
* 분석을 위해 Target을 제외한 4개의 피처 데이터를 표준화해줍니다.
|
1
2
3
4
|
from sklearn.preprocessing import StandardScaler
# Target 값을 제외한 모든 속성 값을 StandardScaler를 이용하여 표준 정규 분포를 가지는 값들로 변환
iris_scaled = StandardScaler().fit_transform(irisDF.iloc[:, :-1])
|
cs |
* pca 라이브러리를 호출하고, 주성분을 2개로 한정해서 모델을 준비합니다.
|
1
2
3
4
5
6
7
8
|
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
#fit( )과 transform( ) 을 호출하여 PCA 변환 데이터 반환
pca.fit(iris_scaled)
iris_pca = pca.transform(iris_scaled)
print(iris_pca.shape)
|
cs |

* 위에서 준비된 모델을 통해 PCA를 한 결과입니다.
|
1
2
3
4
5
|
# PCA 환된 데이터의 컬럼명을 각각 pca_component_1, pca_component_2로 명명
pca_columns=['pca_component_1','pca_component_2']
irisDF_pca = pd.DataFrame(iris_pca, columns=pca_columns)
irisDF_pca['target']=iris.target
irisDF_pca.head(3)
|
cs |

* 이를 바탕으로 시각화해보겠습니다.
|
1
2
3
4
5
6
7
8
9
10
11
12
13
|
#setosa를 세모, versicolor를 네모, virginica를 동그라미로 표시
markers=['^', 's', 'o']
#pca_component_1 을 x축, pc_component_2를 y축으로 scatter plot 수행.
for i, marker in enumerate(markers):
x_axis_data = irisDF_pca[irisDF_pca['target']==i]['pca_component_1']
y_axis_data = irisDF_pca[irisDF_pca['target']==i]['pca_component_2']
plt.scatter(x_axis_data, y_axis_data, marker=marker,label=iris.target_names[i])
plt.legend()
plt.xlabel('pca_component_1')
plt.ylabel('pca_component_2')
plt.show()
|
cs |

앞서 2개의 변수만을 뽑아서 시각화했던 것보다 versicolor와 virginica가 훨씬 잘 구분된 모습을 볼 수 있습니다.
* 주성분별 분산의 설명력은 아래와 같습니다.
|
1
|
print(pca.explained_variance_ratio_)
|
cs |

* 차원을 축소했으니 분류 등 분석간 정확도가 감소 할 수 있습니다. 얼마나 감소하는지 Random Forest를 통해서 알아보겠습니다. 이때, 3fold CV를 해보겠습니다. 먼저 원본 데이터 그대로 사용입니다.
|
1
2
3
4
5
6
7
8
|
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import cross_val_score
import numpy as np
rcf = RandomForestClassifier(random_state=156)
scores = cross_val_score(rcf, iris.data, iris.target,scoring='accuracy',cv=3)
print('원본 데이터 교차 검증 개별 정확도:',scores)
print('원본 데이터 평균 정확도:', np.mean(scores))
|
cs |

* 다음은 PCA 변환 데이터입니다.
|
1
2
3
4
|
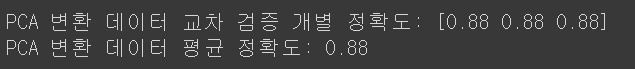
pca_X = irisDF_pca[['pca_component_1', 'pca_component_2']]
scores_pca = cross_val_score(rcf, pca_X, iris.target, scoring='accuracy', cv=3 )
print('PCA 변환 데이터 교차 검증 개별 정확도:',scores_pca)
print('PCA 변환 데이터 평균 정확도:', np.mean(scores_pca))
|
cs |
'머신러닝 with Python' 카테고리의 다른 글
| [Machine Learning] What is machine learning? What is ML? (0) | 2024.03.06 |
|---|---|
| [머신러닝 with 파이썬] 군집화(클러스터링) : K-means & HDBSCAN / 시각화 (0) | 2023.09.27 |
| [딥러닝 with 파이썬] GAN (Generative Adversarial Networks) / 생성적 적대 신경망 / MNIST 데이터로 구현 (2) | 2023.09.25 |
| [머신러닝 with 파이썬] Pycaret이란? Pycaret을 활용한 머신러닝 (0) | 2023.09.24 |
| [머신러닝 with 파이썬] 회귀 트리(Regression Tree) (0) | 2023.09.23 |




댓글