
이번에 알아볼 모델은 DeepLabV3+입니다.
DeepLabV3+는 "Encoder-Decoder with Atrous Seperable Convolution for Semantic Image Segmentation(2018)"이라는 논문에서 나온 모델입니다.
1. DeepLab 모델
- DeepLab은 V1부터 V2, V3, V3+ 까지 발전된 모델인데요. 버전을 거듭하면서 아래와 같은 내용들이 주요 특징이 되겠습니다..
a) DeepLabV1 : Atrous Convolution(Dilated Convolution)을 적용
[개념정리] Dilated Convolution과 Separable Convolution
[개념정리] Dilated Convolution과 Separable Convolution
1. Dilated Convolution - Atrous convolution이라고도 불리는 Dilated convolution은 우리말로 확장 합성곱이라고 불립니다. - 이는 Convolution 커널 내부의 간격을 늘리는 방법으로, 간격을 dilation rate라고 부르며
jaylala.tistory.com
b) DeepLabV2 : 다른 dilation rate을 가진 Atrous conovlution을 한 layer에서 여러개 적용하여 multi-scale context를 적용한 "Atrous spatial pyramid pooling(ASPP)"을 제안
[개념정리] ASPP란? Atrous Spatial Pyramid Pooling 이란?
[개념정리] ASPP란? Atrous Spatial Pyramid Pooling 이란?
이번에 알아볼 것은 Atrous Spatial Pyramid Pooling 입니다. 해당 개념을 이해하기 위해선 Atrous Convolution(Dilated Convolution)에 대한 개념이 선행되어야 하며 이를 위해선 아래 포스팅을 참조하시면 좋습니
jaylala.tistory.com
c) DeepLabV3 : ResNet 구조에 Atrous Convolution을 적용
[딥러닝 with 파이썬] RESNET(잔차신경망)의 개념 (1/2)
[딥러닝 with 파이썬] RESNET(잔차신경망)의 개념 (1/2)
이번에 알아볼 신경망은 잔차 신경망, Residual Net(RESNET) 입니다. 잔차 신경망은 2015년에 처음 등장한 신경망으로, 복잡한 신경망 작업을 원활하게 만들었고 정확도 또한 높게 달성할 수 있었기에
jaylala.tistory.com
d)DeepLabV3+ : Atrous seperable convolution을 제안 ( Depthwise seperable convolution과 Atrous convolution을 결합)
2. DeepLabV3+ 모델의 구조(Architecture)
- 해당 모델의 주요 특징은 아래와 같습니다.
a) 2가지의 인코더를 제시 (DeepLabV3 / 변형된 Xception)
b) ASPP와 Depth-wise Seperable Convolution을 적용
c) Skip architecutre를 활용해서 Encoder와 Decoder가 연결
* Skip-architecture에 대한 설명은 아래 U-Net 논문 리뷰를 통해 확인할 수 있습니다.
[딥러닝 with 파이썬] (논문리뷰)U-Net이란? U-Net: Convolutional Networks for Biomedical Image Segmentation
[딥러닝 with 파이썬] (논문리뷰)U-Net이란? U-Net: Convolutional Networks for Biomedical Image Segmentation
이번에 리뷰할 논문은 U-Net: Convolutional Networks for Biomedical Image Segmentation 입니다. [본 리뷰는 논문 " U-Net: Convolutional Networks for Biomedical Image Segmentation " 와 아래 medium 자료를 참고하여 제작하였습니
jaylala.tistory.com
1) 먼저 DeepLabV3를 Encoder로 한 모델입니다.
* Encoder 부분은 아래와 같으며, ASPP를 적용하였음을 확인할 수 있습니다.
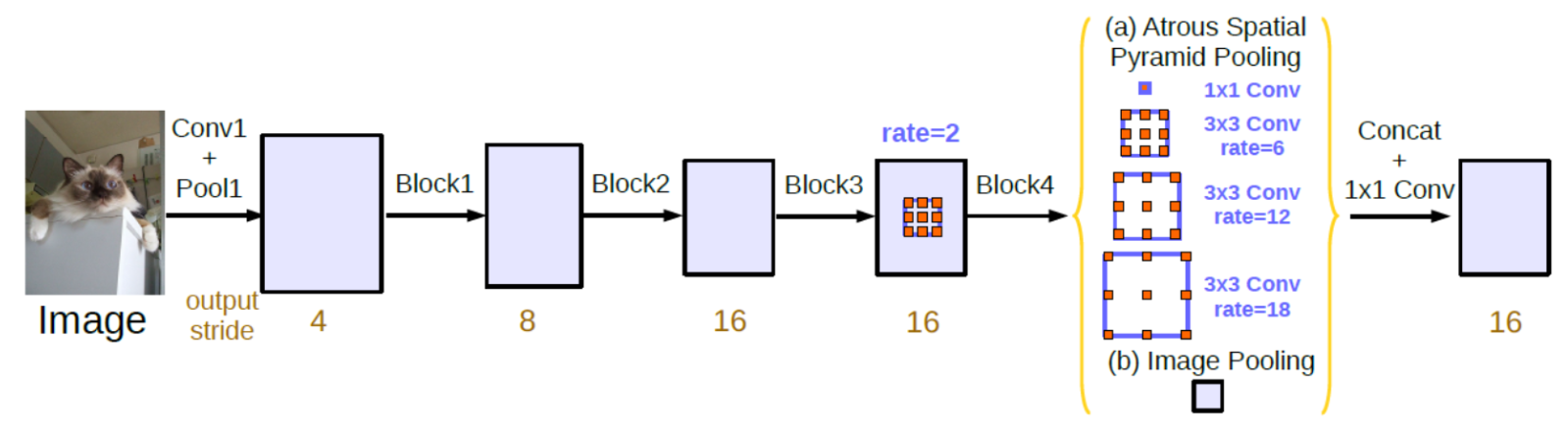
* Decoder 부분은 아래와 같습니다. 기존 DeepLabV3와 Unet을 비교했으며, DeepLabV3의 경우 Bilinear interpolation을 활용하여 Decoding을 합니다. 하지만 이런 방법은 주변 픽셀과의 선형적인 관계를 통해 비어있는 픽셀값이 채워지다보니 Smoothing한 결과가 나와 화질이 떨어지는 결과가 나옵니다. 이를 보완하기 위해, U Net의 Skip Architecture를 활용했으며 모든 Layer에 대해서 적용한 것이 아닌, Bottle Neck 부분과 중간 Layer 부분에서 적용했음을 알 수 있습니다.

* 이를 종합해보면

(1) Encoder에 나온 최종 feature map을 4x bilinear upsampling을 하고
(2) Encoder 중간에 나온 feature map을 1x1 convolution을 적용해 channel을 줄이고
(3) 1번과 2번 feature map을 concatenation 합니다.
(4) 그리고 이를 3x3 convoution을 거쳐 output을 뽑아낸 뒤
(5) 최종 적으로 4x bilinear upsample을 수행해 원래 input size를 복원하여 segemented data를 얻습니다.
2) 변형된 Xception을 Encoder로 한 모델입니다.
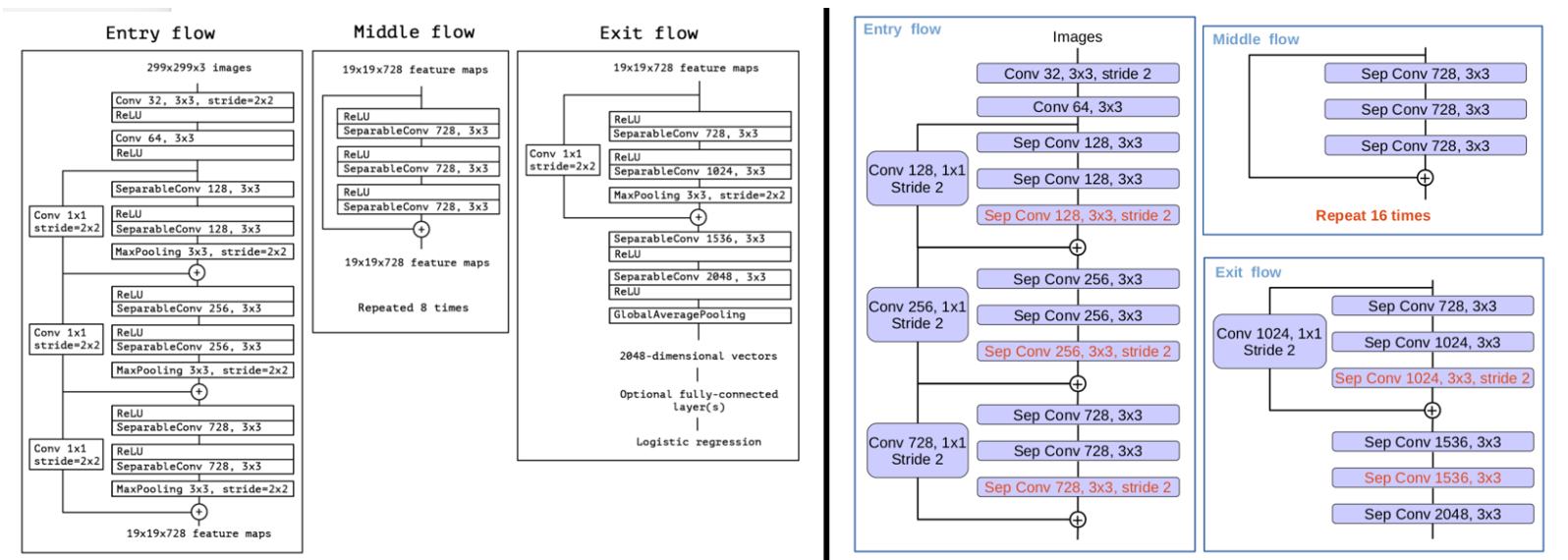
* 좌측이 기존, 우측이 변형된 Xception 모델이며, 더 깊고 Maxpooling을 Depthwise Seperable Convolution을 바꾸었고, 3x3 Convolution 후 Batch Normalization과 ReLU가 연결됨을 확인할 수 있습니다.
성능적인 부분에서
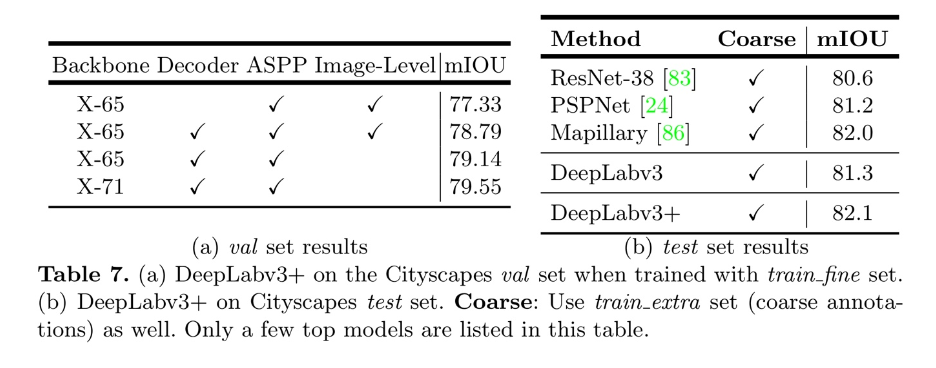
SOTA를 달서했음을 알리고 있습니다. (데이터 : PASCAL VOC 2012, Cityscapes)
'딥러닝 with Python' 카테고리의 다른 글
| [개념정리] Self attention / 셀프 어텐션 (0) | 2024.02.13 |
|---|---|
| [개념정리] 어텐션(Attention)이란 (1) | 2024.02.12 |
| [개념정리] 인공지능에서 임베딩이란 / Embedding (1) | 2024.02.09 |
| [개념정리]Fully Connected CRFs란? (0) | 2024.02.07 |
| [개념정리] ASPP란? Atrous Spatial Pyramid Pooling 이란? (1) | 2024.02.06 |




댓글